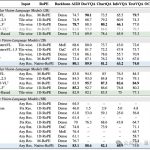
妙啊!用擴散模型生成蛋白質結構,結果不輸天然蛋白質|來自斯坦福&微軟
網友:未來5年用文本提示生成新抗體/新酶也不是夢吧
豐色 發自 凹非寺
量子位 | 公眾號 QbitAI
沒想到,圖像生成領域的大明星——
擴散模型,這么快就被用來做蛋白質結構生成了!
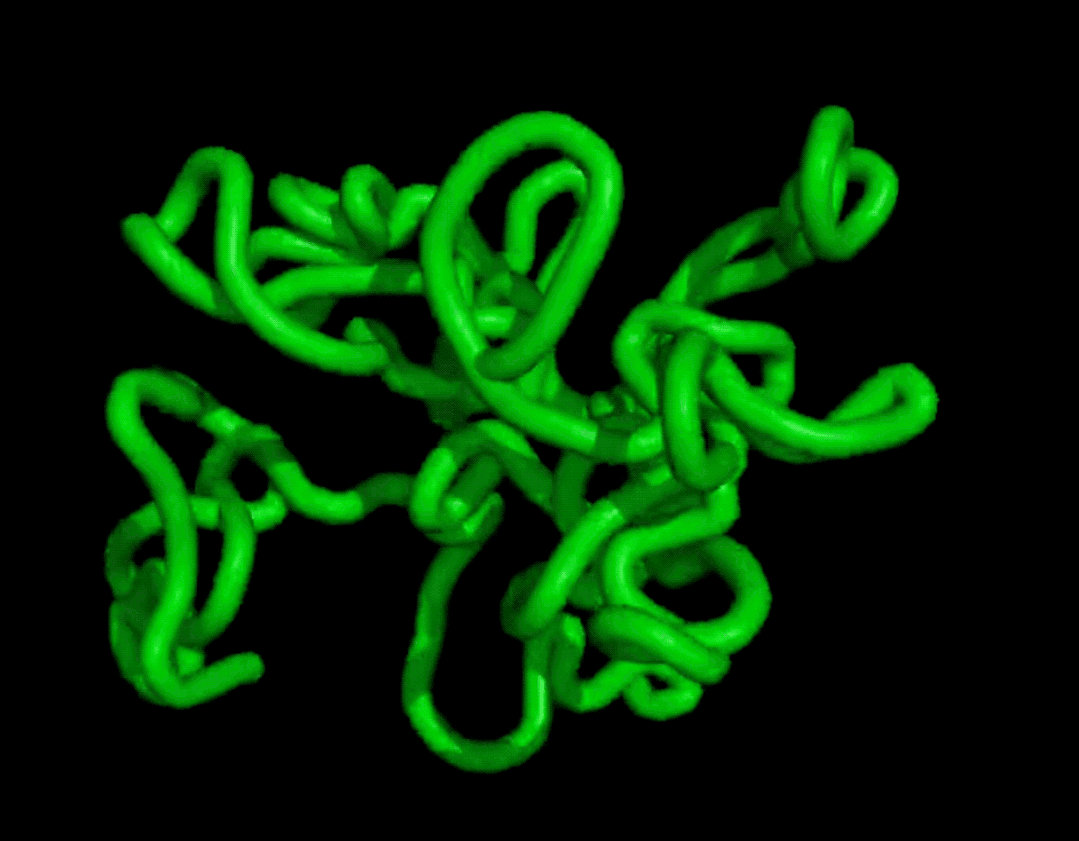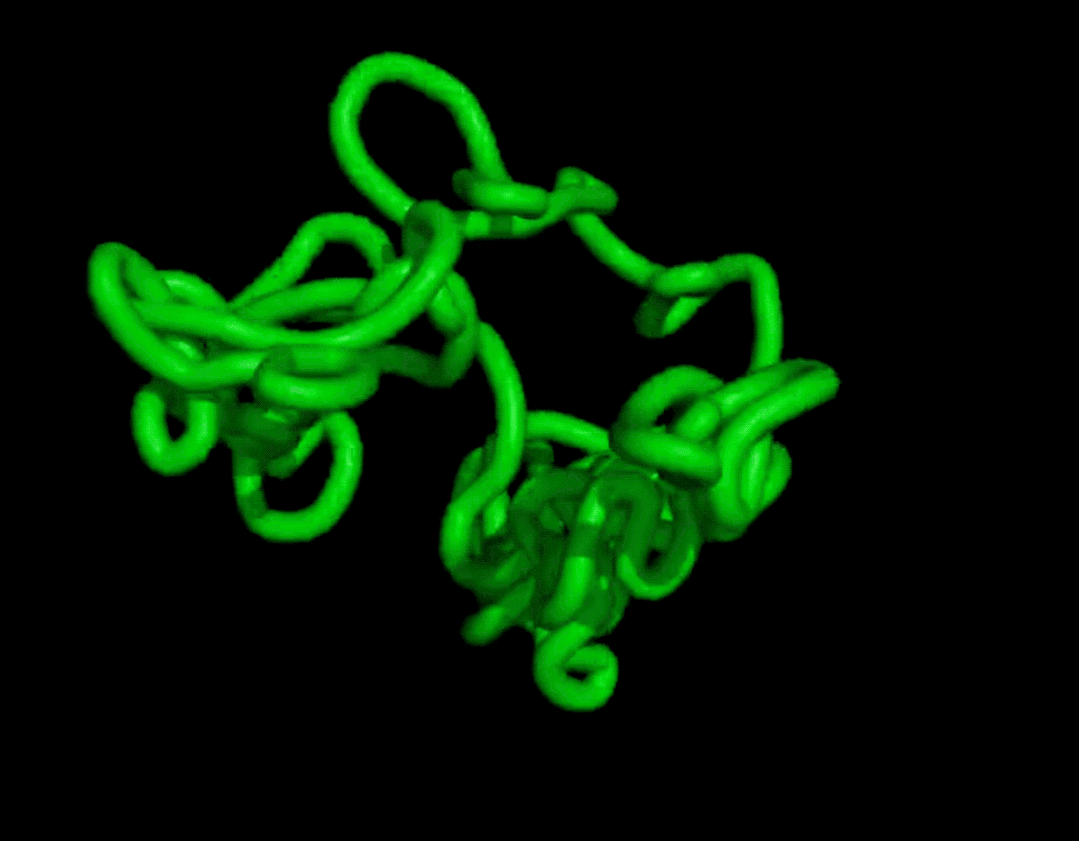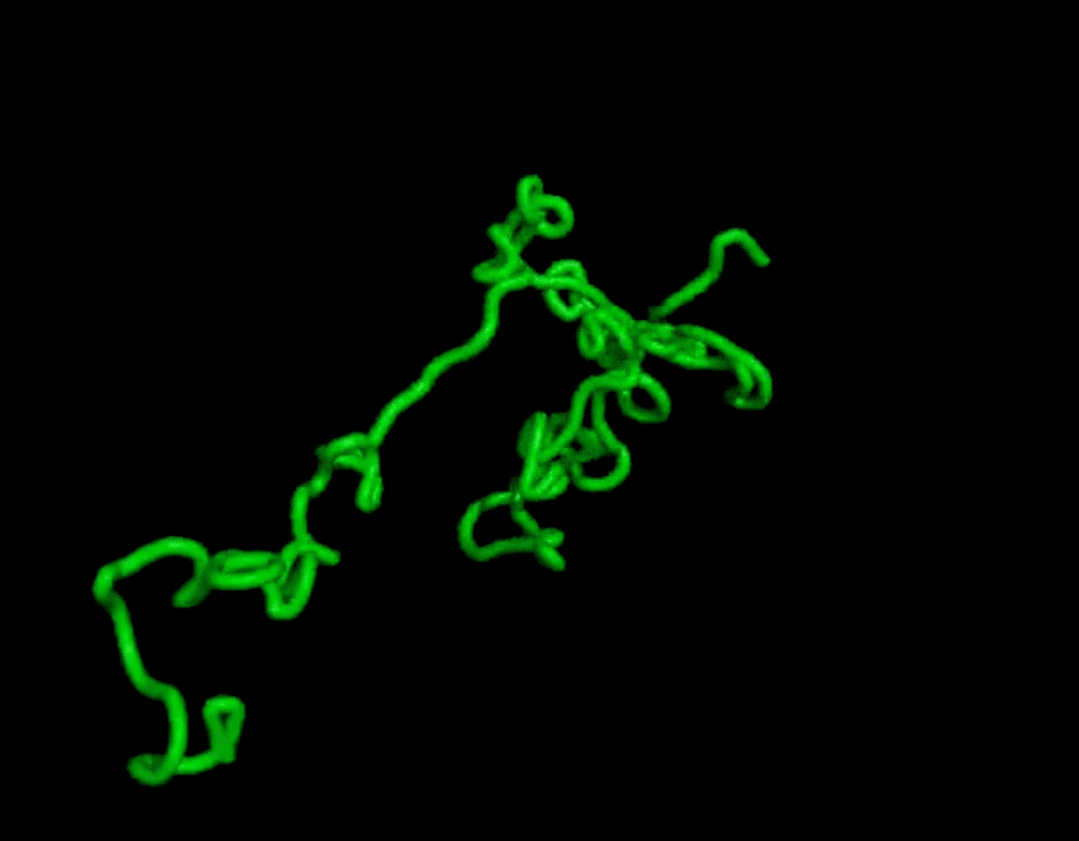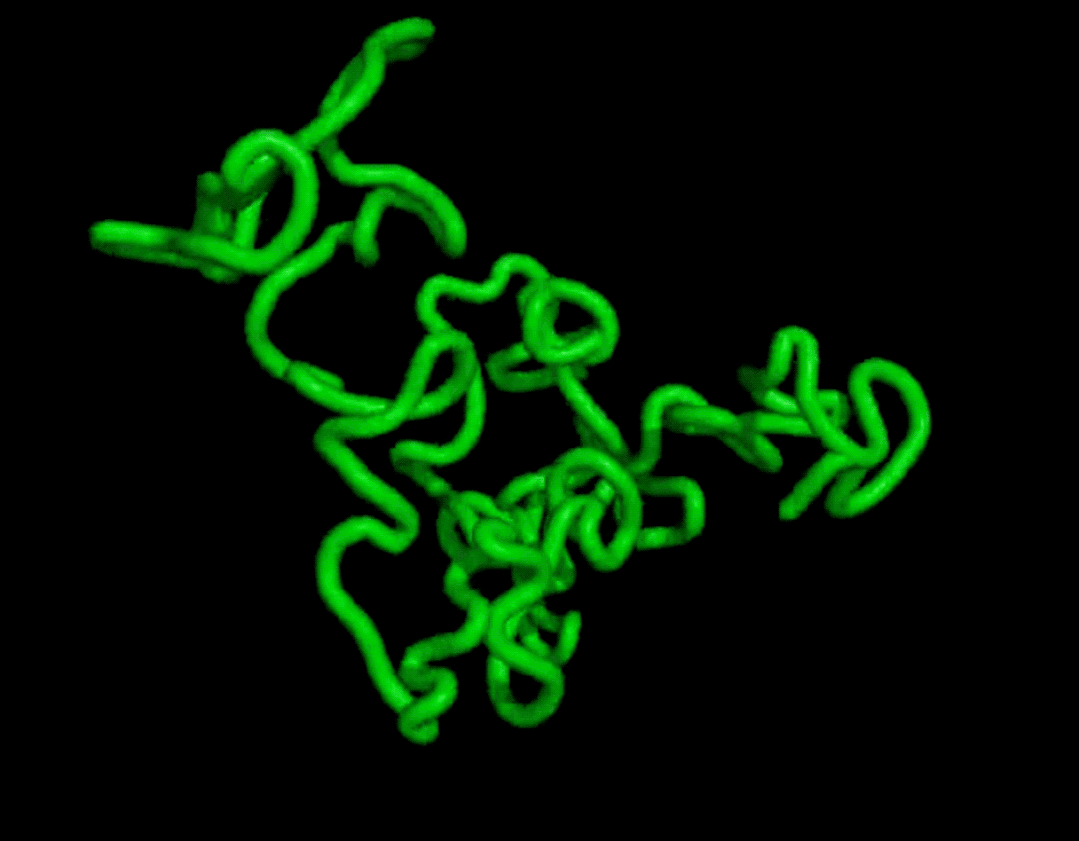
而且結果在復雜度和結構上都和天然蛋白質有的一拼。

消息一出,不少人都稱贊這個組合簡直非常妙。

還有人表示:我早就猜到了,生成模型能做的真的不僅是圖像和視頻。
所以,AlphaFold這是可能有新的挑戰者了?

具體是怎么回事?
來看看斯坦福大學和微軟的這項最新研究成果到底怎么說。

擴散模型vs蛋白質結構生成
說起研究的初衷,作者表示:
盡管蛋白質結構預測已經取得了非常好的成績,但要從神經網絡中直接生成多結構多樣又新穎的蛋白質結構仍然很困難。
他們想到用基于擴散的生成模型來挑戰這一任務,并通過鏡像蛋白質自然折疊過程來設計蛋白質主鏈結構。
具體來說,就是將蛋白質主鏈結構看成一系列連續的角度,這些角度會捕捉組成氨基酸殘基的相對方向。
進而通過從隨機、未折疊狀態到穩定折疊結構的去噪就可以生成新結構。
作者表示,這一設計不僅可以反映蛋白質如何在生物學上扭曲成能量上有利的結構(how proteins biologically twist into energetically favorable conformations),這種表示的固有位移和旋轉不變性也可以極大地減輕模型對復雜等變網絡的需要。
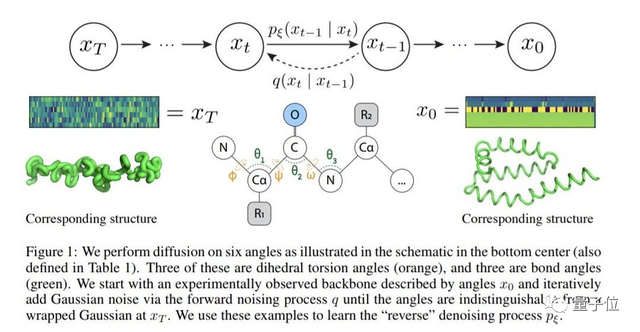
在實現上,作者僅用一個簡單的transformer作為backbone就訓練出了一個去噪擴散概率模型。
最終證明它可以無條件地生成高度真實的蛋白質結構,其復雜性和結構模式類似于天然蛋白質的結構模式。
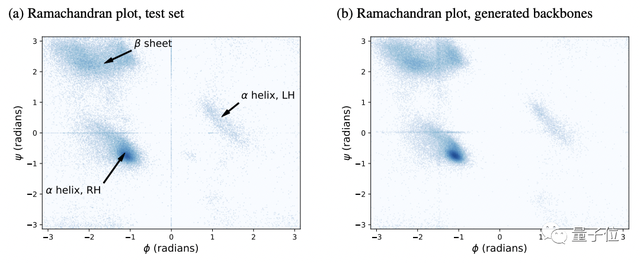
如下圖Ramachandran plot(拉氏圖,專門用于檢測蛋白質構象是否合理)所示,左右分別為測試集和生成的蛋白質主干的(φ,ψ)二面角。
可以看到,三個主要結構元素、以及一些不太常見的角度組合都在他們用擴散模型生成的主干中得到了呈現。
目前,基于以上成果,作者已公開發布了首個用于蛋白質結構擴散的開源代碼庫和訓練模型,詳情可戳文末鏈接。
不過,作為一個初步探索,他們也指明這項成果還存在幾個局限性,比如:
1、與通常有幾百個殘基的天然蛋白質相比,模型生成的結構仍然相對較短(最多128個殘基);
2、由于沒有處理多鏈復合物或配體相互作用,模型無法捕獲蛋白質的動態性質,只能生成靜態結構;
3、將蛋白質表述為一系列角度的框架設計會造成一些累積誤差,最終顯著改變生成的整體結構。
最后一個問題則可以對未來工作提供思路,比如試著用幾何信息架構中使用的方法來解決相關問題。
最后,除了再次刷新我們對擴散模型的認知,一位畢業于ETH的研究人員看完這項成果后還大膽預測道:
未來5年內,可能漸漸沒有人會對全新的蛋白質序列或折疊感到興奮了。
因為潛在的新酶和治療性抗體將通過文本提示生成。

對于這項成果,你有什么想說的?
論文地址:
https://arxiv.org/abs/2209.15611
代碼:
https://github.com/microsoft/foldingdiff
- 北大開源最強aiXcoder-7B代碼大模型!聚焦真實開發場景,專為企業私有部署設計2024-04-09
- 剛剛,圖靈獎揭曉!史上首位數學和計算機最高獎“雙料王”出現了2024-04-10
- 8.3K Stars!《多模態大語言模型綜述》重大升級2024-04-10
- 谷歌最強大模型免費開放了!長音頻理解功能獨一份,100萬上下文敞開用2024-04-10